Transmissionselektronenmikroskopie (TEM) und Rastertransmissionselektronenmikroskopie (STEM)
Mit der Transmissionselektronenmikroskopie (TEM) und Rastertransmissionelektronenmikroskopie (STEM) wird die projizierte Struktur von dünnen, elektronentransparenten Proben abgebildet. Durch Kombination mit einem HAADF (high-angle annular dark-field) Detektor können mit der STEM chemisch sensitive Abbildungen erzeugt werden. Die bestmögliche Auflösung liegt im Bereich von 0.1 nm. Voraussetzung für TEM Untersuchungen ist, dass aus dem Ausgangsmaterial eine elektronentransparenter Probe präpariert werden kann. Die maximal durchstrahlbare Probendicke liegt in der Größenordnung von 1000 nm. Für hochauflösende TEM Abbildungen sind Probendicken unter 50 nm erforderlich. Zur Präparation materialwissenschaftlicher Proben sind alle notwendigen Einrichtungen einschließlich eines Focused-Ion-Beam (FIB) Systems vorhanden. Biologische Dünnschnitte können mit einem Ultramikrotom hergestellt werden. Im Beugungsmodus können lokale Beugungsbilder (Feinbereichsbeugung) aufgenommen werden, anhand derer Gitterebenenabstädne und Kristallstrukturen ermittelt werden können.
Die TEM kann in Kombination mit analytischen Technken (energiedispersive Röntgenspektroskopie (EDXS), Elektronenenergieverlustspektroskopie (EELS) und energiefilternder TEM (EFTEM)) für chemische Analysen, zur Untersuchung von Bindung- und Oxidationszuständen sowie der elektronischen Eigenschaften eingesetzt werden. Mit einem auf kleinen Durchmesser fokussierten Elektronenstrahl können die analytischen Untersuchungen mit sub-Nanometer Auflösung durchgeführt werden.
Elektronenholographie im Transmissionselektronenmikroskop erlaubt die Rekonstruktion der Wellenfunktion an der Probenunterseite. Die Phase der Wellenfunktion enthält u.a. Information über innere und äußere elektrische und magnetische Felder. Tomographische Untersuchungen liefern Informationen über die dreidimensionale Struktur von Nanoteilchen, Ausscheidungen und Poren.
Typische Einsatzgebiete der TEM sind:
-
Charakterisierung von Defekten (Versetzungen, Stapelfehler), Bestimmung der Verteilung und Dichte der Defekte
-
atomare Struktur von Grenzflächen, Nanoteilchen, .....
-
Verteilung und Größe von Ausscheidungen, Partikeln und Hohlräumen
-
Analyse unterschiedlicher Phasen, Darstellung der Verteilung unterschiedlicher Elemente
-
Korngrenzenstruktur und Segregation von Fremdatomen in Korngrenzen
-
Abbildung innerer nanoskaliger elektrischer Felder, z.B. an geladenen Versetzungen und Korngrenze
Ansprechpartner:
Beispiele
Querschnittsabbildung von nanokristallinen (LaSr)CoO3 Schichten auf Y-dotiertem ZrO2 und Gd-dotiertem CeO2: farbkodiert dargestellte Verteilung von La, Sr, Co, Zr
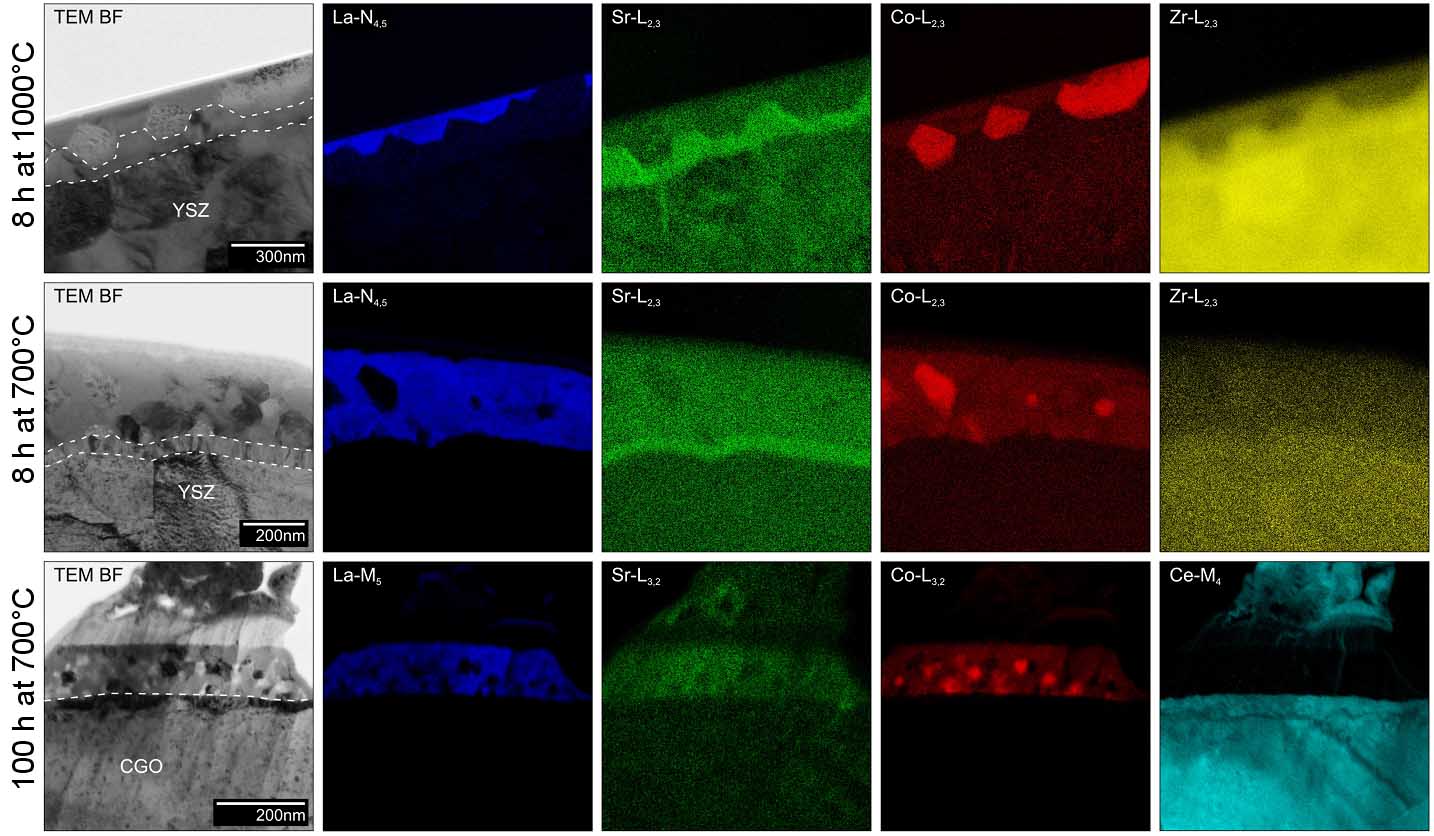
L. Dieterle et al., J. Mat. sci. 43, 3135 (2008)
Pt-Cluster auf einer dünnen amorphen Kohlenstoffschicht

Reinhard Schneider, Holger Blank (Laboratorium für Elektronenmikroskopie)
Querschnittsabbildungen einer Zn0.69Mn0.31Se/GaAs(001) Halbleiter-Heterostruktur: (a) Beugungsbild entlang der [110]-Zonenachse mit Zwillingsorientierungen (2,3) und (b) Dunkelfeldabbildung vom Bereich des Beugungsbildes mit Abbildungsvektor g=(220), (c) Dunkelfeldabbildung mit g=(111).

D. Litvinov et al., J. Appl. Phys.100, 023523 (2006)
Querschnittsabbildung einer GaAs/Si(001) Halbleiter-Heterostruktur: Versetzungen in einer epitaktischen GaAs Schicht auf Silizium Substrat

D. Gerthsen (Laboratorium für Elektronenmikroskopie)
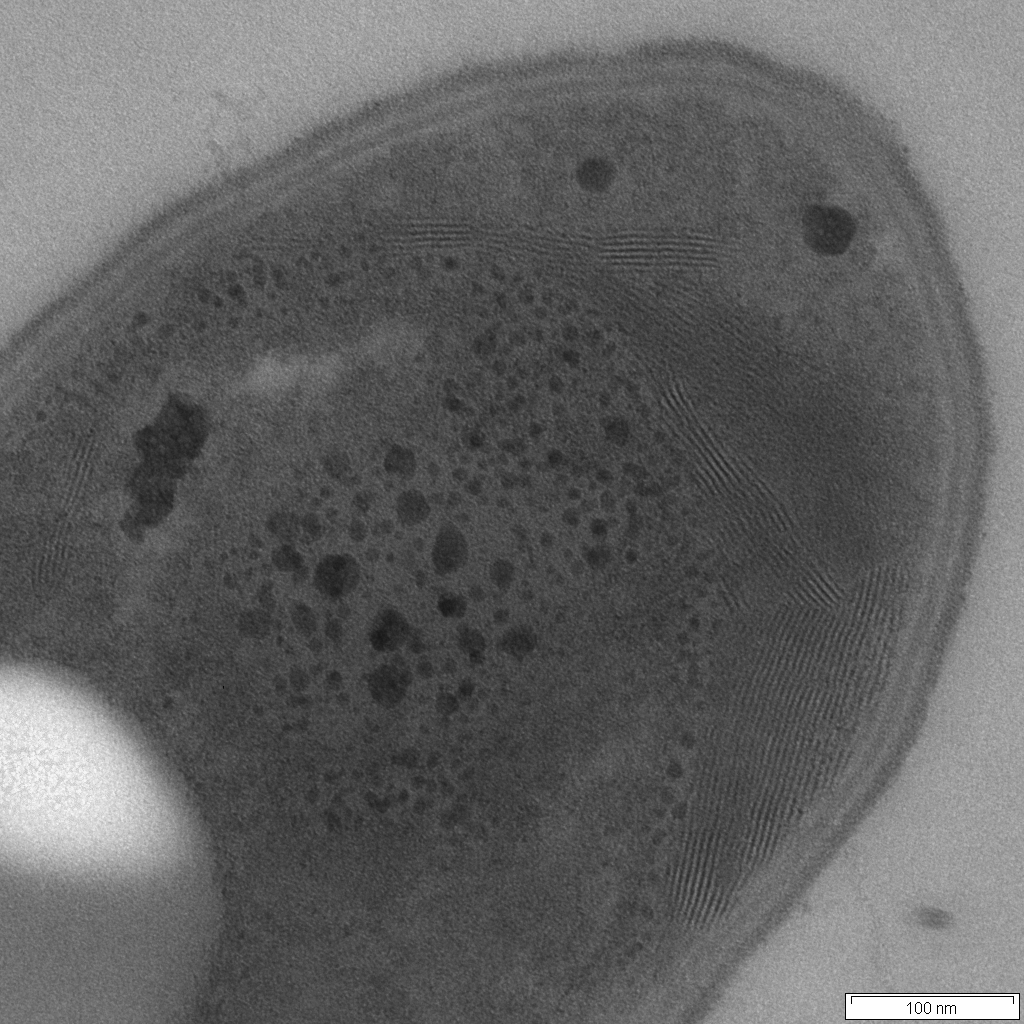
Bakterien
Dense packing lamellar oriented membrane structures and multiple electron dense inclusion bodies on apical part of bacterial cells of Aneurinibacillus migulanus DSM 5658, producing Gramicidin S. Ultrathin section of cells, taken during the transition to stationary growth phase, were done by Dr. Marina Berditsch (KIT, Institute of Organic Chemistry, Department Biochemistry) and TEM by M. Fotouhi (Laboratory for Electron Microscopy).